Structuurbiologie
| Deel van een serie artikelen over Biochemie | ||||
|---|---|---|---|---|
| ||||
| Ruimtelijke structuur van cytochroom c | ||||
| Biomoleculen | ||||
|
Eiwit · Koolhydraat · Natuurproduct · | ||||
| Stofwisseling | ||||
|
Anabolisme · Celademhaling · Eiwitsynthese · Katalyse · Fotosynthese · Katabolisme | ||||
| Verwante onderwerpen | ||||
| ||||
Structuurbiologie (Engels: structural biology) is een deelgebied van de biochemie waarin men de ruimtelijke structuur bestudeert van biologische macromoleculen (met name eiwitten en nucleïnezuren), hoe zij zich in deze structuur organiseren en hoe veranderingen in hun structuur hun biochemische functie beïnvloeden.
Eiwitten en nucleïnezuren liggen aan de basis van vrijwel alle cellulaire processen, en enkel de specifieke driedimensionale structuur van dergelijke macromolecuul bepaalt hoe deze functies worden uitgevoerd. De ruimtelijke architectuur van macromoleculen is vaak terug te voeren op de volgorde (sequentie) van de monomeren waaruit ze zijn opgebouwd. Eiwitten vouwen zich bijvoorbeeld op in een complexe tertiaire structuur, op basis van hun aminozuursequentie, de primaire structuur.
Structuurbiologisch onderzoek wordt gedaan op basis van technieken uit de biofysica en biochemie. De meeste biomoleculen zijn te klein om met microscopen in detail waar te nemen. Een aantal belangrijke technieken die structuurbiologen gebruiken zijn massaspectrometrie, röntgendiffractie, kernspinresonantie (NMR) en small-angle X-ray scattering. De ontwikkeling van cryo-elektronenmicroscopie, een vorm van atomaire microscopie, stelde onderzoekers in staat de macromoleculaire structuur van eiwitten of andere celcomponenten zichtbaar te maken in hun natuurlijke ruimtelijke staat.
Ook principes uit de bio-informatica kunnen worden gebruikt om structuren te onderzoeken, door naar verbanden te kijken tussen de vouwingsmotieven in de nucleotidesequentie van het macromolecuul. Voorspelling van eiwitstructuren met behulp van AI-tools zoals AlphaFold zijn vanaf 2018 steeds krachtiger en accurater geworden, en nemen een steeds belangrijkere plaats in in de structuurbiologie. Het ontwerpen van eiwitten met gewenste eigenschappen (protein engineering) is mede hierdoor in een stroomversnelling gekomen.
Geschiedenis
In 1912 experimenteerde de natuurkundige Max von Laue met het richten van röntgenstraling op gekristalliseerde materialen, wat leidde tot de ontdekking van diffractiepatronen. Dit markeerde het begin van de röntgendiffractie en uiteindelijk de ontwikkeling van de röntgenkristallografie, een techniek die later essentieel werd voor het bestuderen van biologische moleculen. In 1951 gebruikten Rosalind Franklin en Maurice Wilkins röntgendiffractie om de eerste beelden van DNA vast te leggen. Op basis van deze gegevens modelleerden James Watson en Francis Crick in 1953 de dubbele helixstructuur van DNA. Samen met Wilkins ontvingen zij hiervoor in 1962 de Nobelprijs voor de Geneeskunde.
Het eerste eiwit dat succesvol gekristalliseerd werd voor röntgendiffractie was pepsine, door Theodore Svedberg, die in 1962 de Nobelprijs voor de Chemie ontving. In 1958 werd de eerste driedimensionale structuur van een eiwit (myoglobine) gepubliceerd door John Kendrew. Destijds werden eiwitstructuren nog met houten of metalen modellen nagebouwd.[1] Rond 1980 werd hiervoor computermodellen gebruikt, bijvoorbeeld via het softwarepakket CCP4.
In de late jaren 1930 en vroege jaren 1940 leidde werk van Isidor Rabi, Felix Bloch en Edward Purcell tot de ontwikkeling van kernspinresonantie (NMR), een techniek die tegenwoordig breed wordt ingezet voor het analyseren van eiwitstructuur en eiwitdynamiek. NMR kon atomaire details onthullen van eiwitten in oplossing, inclusief dynamiek en interacties, zonder dat kristallisatie vereist was.
Tenslotte zijn computationele methoden steeds belangrijker geworden. Moleculaire dynamica (MD) wordt gebruikt om de beweging van moleculen te simuleren. In 1975 werd de eerste eiwitvouwing met MD gepubliceerd. Vanaf 2018 heeft AlphaFold, een deep learning-model, een grote doorbraak betekend in het voorspellen van eiwitstructuren. Sommigen beschouwen computationele technieken en AI-tools inmiddels als leidend binnen het veld van de structuurbiologie.[2]
-
.jpg) Rosalind Franklin, DNA-structuur via röntgendiffractie
Rosalind Franklin, DNA-structuur via röntgendiffractie -
 John Kendrew, eerste eiwitstructuur (myoglobine)
John Kendrew, eerste eiwitstructuur (myoglobine) -
 Isidor Rabi, grondlegger van NMR-techniek
Isidor Rabi, grondlegger van NMR-techniek
Toepassingen
Structuurbiologie heeft bijdragen aan de ontrafeling van de moleculaire mechanismen die ten grondslag liggen aan vele ziekten. NMR en cryo-EM zijn bijvoorbeeld gebruikt om de aggregatie van amyloïde-eiwitten te bestuderen, die een rol spelen in de ziekte van Alzheimer, de ziekte van Parkinson en diabetes type II.[3] Kennis van de vouwingsstructuur van deze eiwitten zijn cruciaal voor begrip van het ziektemechanisme. Structuurbiologische technieken worden bovendien ingezet om interacties tussen ziekteverwekkers en hun gastheer te verklaren. Zo zijn deze technieken ingezet om het envelop-eiwit van HIV te bepalen, waardoor onderzoekers nu beter begrijpen hoe dit virus het menselijk immuunsysteem ontwijkt.
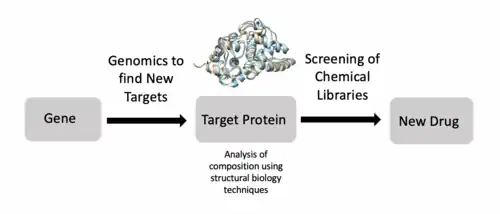
Structuurbiologie speelt ook een belangrijke rol in het geneesmiddelonderzoek.[4] Wetenschappers kunnen via genomica eiwitten vinden die mogelijk ten grondslag liggen aan een ziekte, en deze doel-eiwitten vervolgens bestuderen middels structuuranalyse, om zo geneesmiddelen te ontwikkelen die specifiek op deze doelwitten zijn afgestemd. Zo hebben onderzoekers de structuur van het Met-eiwit opgehelderd, een receptor-tyrosinekinase die een rol kan spelen bij de groei en verspreiding van kankercellen, met name bij long- en maagkanker. Door de bindingsplaatsen van Met in kaart te brengen, konden farmaceuten doelgerichte remmers ontwikkelen zoals crizotinib, die de activiteit van het eiwit blokkeren.[4] Verder worden bij de bestrijding van mycobacteriële infecties, zoals tuberculose, nieuwe antibiotica ontwikkeld op basis van de 3D-structuren van essentiële enzymen van Mycobacterium tuberculosis, bijvoorbeeld door te richten op het enzym InhA, dat betrokken is bij vetzuursynthese in de bacteriële celwand.[4]
Zie ook
Referenties
- ↑ (en) Garman EF (2014). Developments in x-ray crystallographic structure determination of biological macromolecules. Science 343 (6175): 1102–1108. PMID 24604194. DOI: 10.1126/science.1247829.
- ↑ (en) Nussinov R, Tsai CJ, Shehu A, Jang H. (2019). Computational Structural Biology: Successes, Future Directions, and Challenges. Molecules 24 (3): E637. PMID 30759724. PMC 6384756. DOI: 10.3390/molecules24030637.
- ↑ (en) Fitzpatrick AW, Falcon B, He S, Murzin AG, Scheres S. (2017). Cryo-EM structures of tau filaments from Alzheimer's disease. Nature 547 (7662): 185–190. PMID 28678775. DOI: 10.1038/nature23002.
- ↑ a b c (en) Thomas SE, Mendes V, Kim SY, Malhotra S, Ochoa-Montaño B, Blaszczyk M, Blundell TL (2017). Structural Biology and the Design of New Therapeutics: From HIV and Cancer to Mycobacterial Infections: A Paper Dedicated to John Kendrew. Journal of Molecular Biology John Kendrew’s 100th Anniversary Special Edition 429 (17): 2677–2693. PMID 28648615. DOI: 10.1016/j.jmb.2017.06.014.
Literatuur
- (en) Banaszak, LJ. (2000). Foundations of Structural Biology. Elsevier Academic Press, Burlington. ISBN 978-0-08-052184-8.